Métodos de Análise de Cluster: diana, daisy e pam
Multivariada
Cluster
Introdução
Você não precisa usar o R… pode usar o Python também.
Análise de Cluster
O nome Cluster contém um amplo conjunto de técnicas para localizar subgrupos de observações em um banco de dados. Quando agrupamos observações, queremos que as observações no mesmo grupo sejam semelhantes e as observações em grupos diferentes sejam diferentes. Como não há uma variável resposta, esse é um método não supervisionado. O agrupamento nos permite identificar quais observações são semelhantes e potencialmente categorizá-las.
Existem diferentes tipos de métodos de cluster. hoje vamos discutir como fazer:
- Cluster hierárquico
- Cluster via K-means e K-means++
- Cluster fuzzy
- Cluster para variaveis categóricas
- Cluster PAM
- Density-based clustering
- t-SNE plot
Obs- essa lista não representa uma ordem.
No total, há três decisões (relacionadas entre si) que precisam acontecer em um método de análise de cluster:
- Cálculo de Distância.
- Escolher um algoritmo de cluster.
- Selecionando o número de clusteres.
Preparação do banco de dados
Para executar uma análise de cluster no R,o banco de dados devem ser preparados da seguinte maneira:
- As linhas são observações (indivíduos) e colunas são as variáveis.
- Qualquer valor ausente (missing) nos dados deve ser removido ou estimado.
- Os dados devem ser padronizados (ou seja, dimensionados) para tornar variáveis comparáveis. Lembre-se de que a padronização consiste em transformar as variáveis de forma que elas tenham média zero e desvio padrão igual a um.
Cluster hierárquico
De acordo com https://uc-r.github.io/hc_clustering, o agrupamento hierárquico pode ser dividido em dois tipos principais: AGNES e DIANA.
- AGNES (agrupamento aglomerado). Funciona de maneira ascendente. Ou seja, cada objeto é inicialmente considerado como um cluster de elemento único. Em cada etapa do algoritmo, os dois clusters mais semelhantes são combinados em um novo cluster maior (nós). Este procedimento é iterado até que todos os pontos sejam membros de apenas um único grande cluster (raiz). O resultado é uma árvore chamada dendograma.
- DIANA. Funciona de maneira descendente. O algoritmo é uma ordem inversa de AGNES. Começa com a raiz, na qual todos os objetos são incluídos em um único cluster. Em cada etapa da iteração, o cluster mais heterogêneo é dividido em dois. O processo é iterado até que todos os objetos estejam em seu próprio cluster.
#----------------------------------------------
# Hierarquico
#----------------------------------------------
library(cluster) # clustering algorithms
library(factoextra) # clustering visualization
library(dendextend) # for comparing two dendrograms
# To standarize the variables
wine.stand <- scale(wine[-1])
# Dissimilarity matrix
d <- dist(na.omit(wine.stand), method = "euclidean")
# Hierarchical clustering using Complete Linkage
hc1 <- hclust(d, method = "complete" )
plot(hc1, cex = 0.6, hang = -1)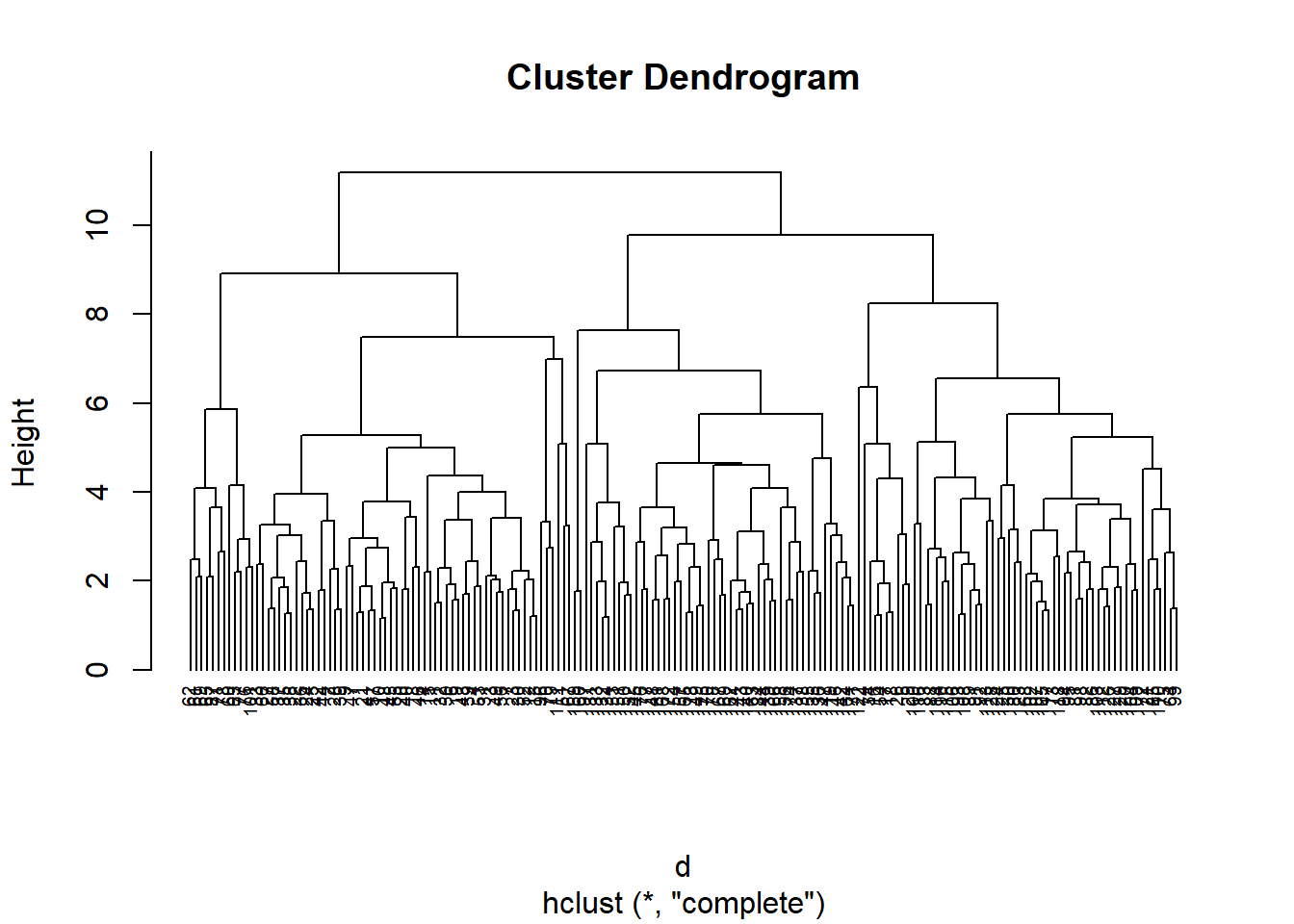
#Agglomerative Nesting (Hierarchical Clustering)
hc2 <- agnes(na.omit(wine.stand), method = "ward")
pltree(hc2, cex = 0.6, hang = -1, main = "Dendrograma") 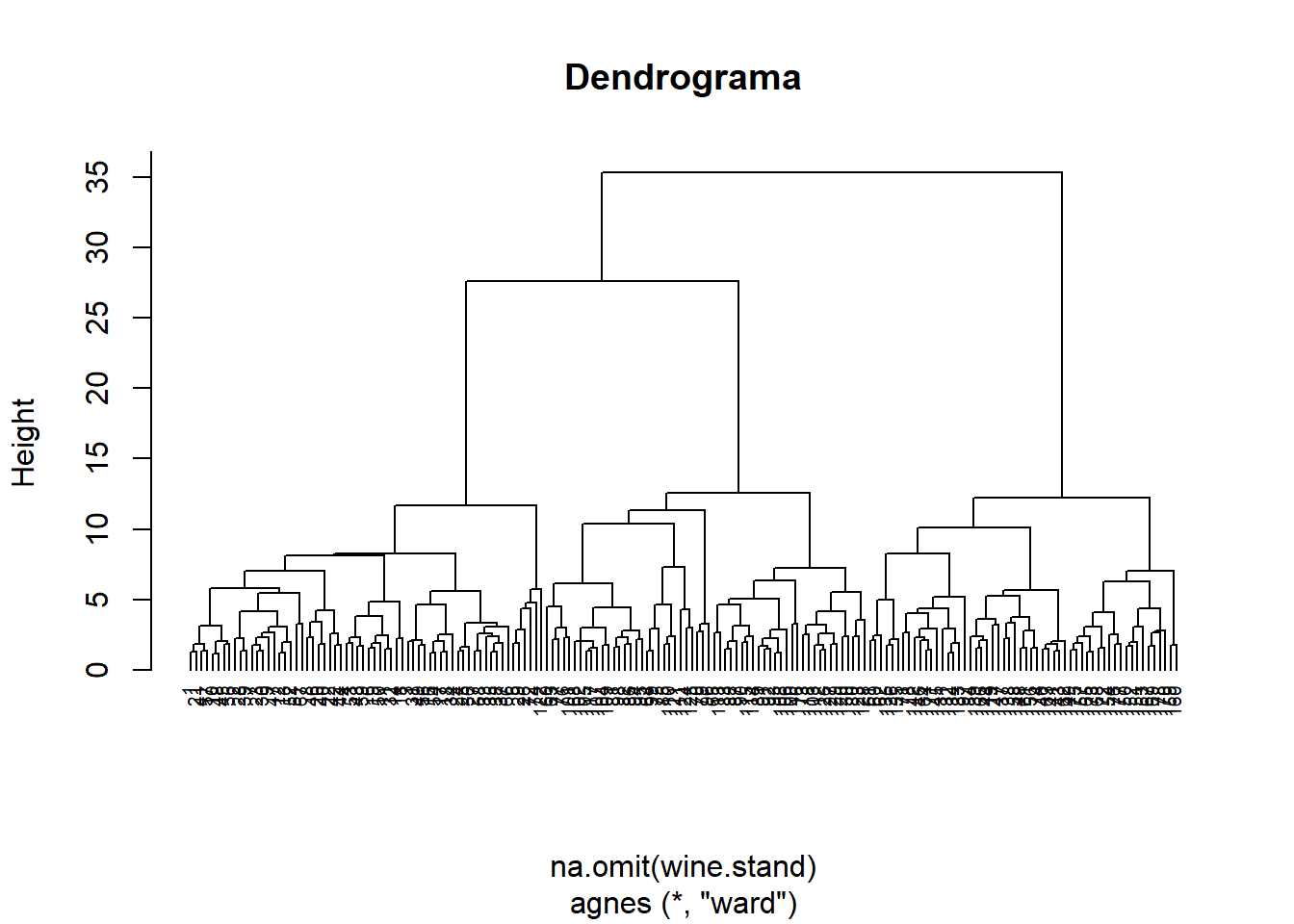
# DIvisive ANAlysis Clustering - DIANA
hc3 <- diana(na.omit(wine.stand),metric = "manhattan")
# Cut tree into 5 groups
tres_gruspos <- cutree(as.hclust(hc3), k = 3)
#dados_completos<-merge(wine.stand,tres_grupos,by="row.names",all.x=TRUE)Correlação Cofonética no R
A distância cofenética entre duas observações que foram agrupadas é definida como a dissimilaridade intergrupos na qual as duas observações são combinadas primeiro em um único agrupamento.
Pode-se argumentar que um dendrograma é um resumo apropriado de dados se a correlação entre as distâncias originais e as distâncias copenéticas for alta.
corr_coph<-cophenetic(hc1)
#corr_cophCluster K-means e K-means-plus-plus
O Cluster K-means é o método de agrupamento mais simples e mais usado para dividir um conjunto de dados em um conjunto de k grupos.
# K-Means
k_means_2 <- kmeans(wine.stand, 2) # k = 2
attributes(k_means_2)$names
[1] "cluster" "centers" "totss" "withinss" "tot.withinss"
[6] "betweenss" "size" "iter" "ifault"
$class
[1] "kmeans"# Centroids:
k_means_2$centers Alcohol Malic Ash Alcalinity Magnesium Phenols
1 0.04186090 -0.3811653 -0.1089132 -0.2963363 0.08872942 0.5413132
2 -0.07277357 0.6626412 0.1893414 0.5151693 -0.15425269 -0.9410522
Flavanoids Nonflavanoids Proanthocyanins Color Hue Dilution
1 0.6003536 -0.4806503 0.4107892 -0.3117353 0.5059593 0.6133643
2 -1.0436916 0.8355920 -0.7141412 0.5419399 -0.8795908 -1.0663102
Proline
1 0.2599460
2 -0.4519062# Clusters:
k_means_2$cluster [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
[38] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 1 1 1 1 1 1 2 1 2 1 1 1
[75] 1 1 1 2 1 1 1 1 1 2 1 1 1 1 2 1 2 2 2 1 1 1 1 1 1 1 1 1 1 1 1 2 1 2 1 1 1
[112] 1 2 1 1 1 1 1 2 1 1 1 2 1 1 1 1 2 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
[149] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2# Cluster size:
k_means_2$size[1] 113 65# Visualização
fviz_cluster(k_means_2, data = wine.stand)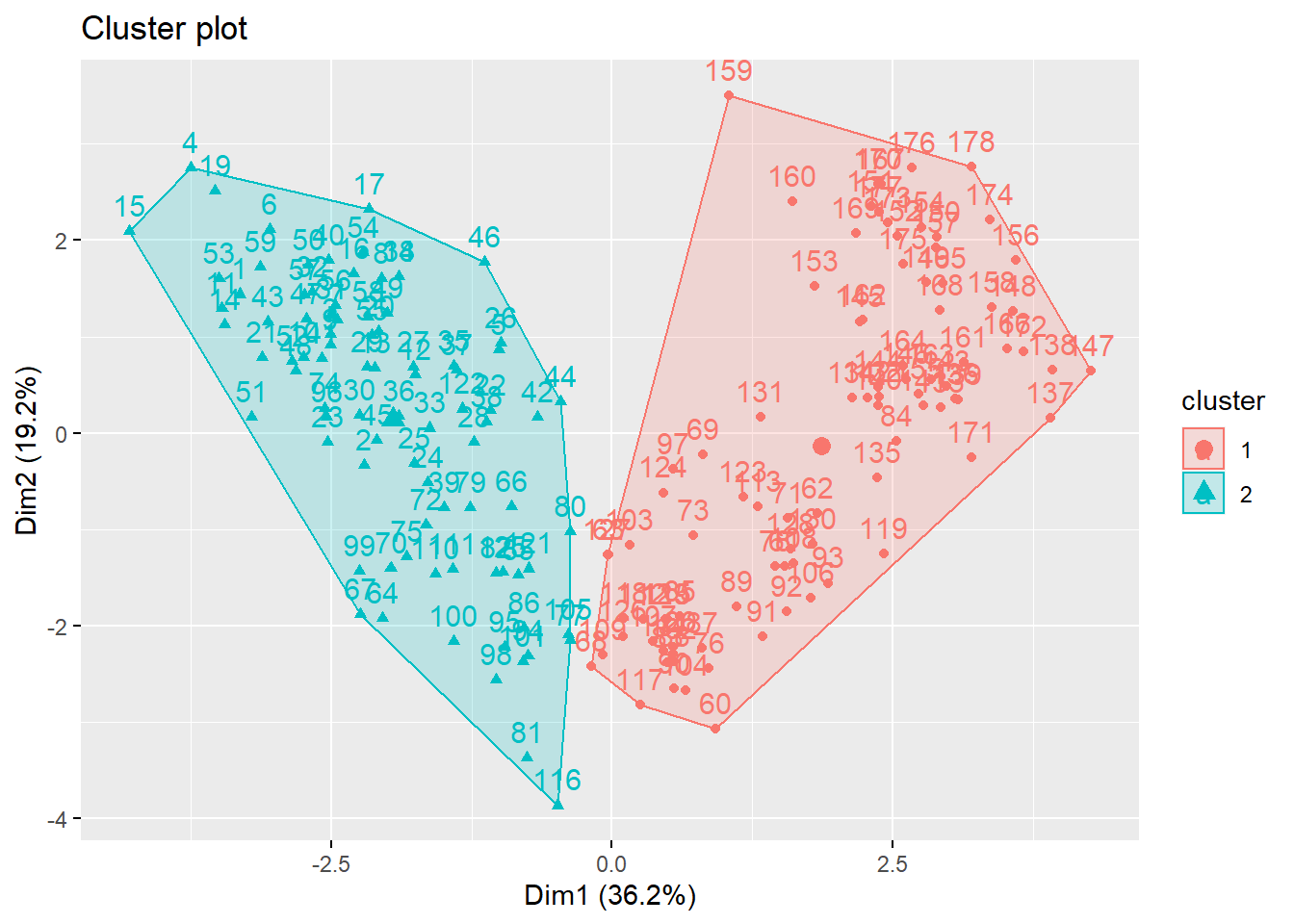
Podemos fazer uma análise de sensibilidade com k=3,4, e 5.
k_means_3 <- kmeans(wine.stand, centers = 3, nstart = 25)
k_means_4 <- kmeans(wine.stand, centers = 4, nstart = 25)
k_means_5 <- kmeans(wine.stand, centers = 5, nstart = 25)
# plots to compare
p1<-fviz_cluster(k_means_2, geom = "point", data = wine.stand) + ggtitle("k = 2")
p2<-fviz_cluster(k_means_3, geom = "point", data = wine.stand) + ggtitle("k = 3")
p3<-fviz_cluster(k_means_4, geom = "point", data = wine.stand) + ggtitle("k = 4")
p4<-fviz_cluster(k_means_5, geom = "point", data = wine.stand) + ggtitle("k = 5")
library(gridExtra)
grid.arrange(p1, p2, p3, p4, nrow = 2)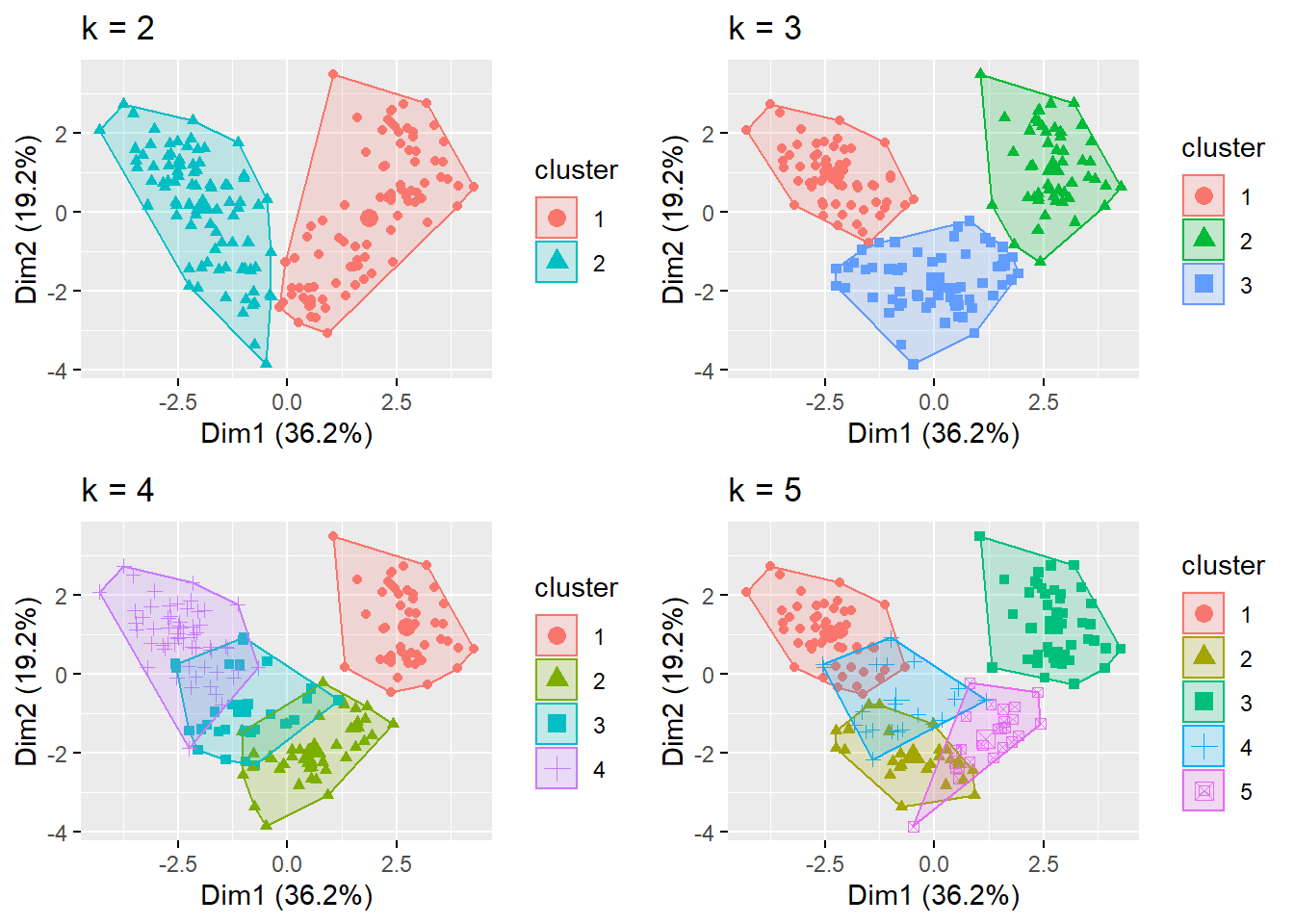
k-means++ é um algoritmo para a escolha dos valores iniciais (ou “sementes”) para o tradicional algoritmo de cluster k-means.
library(clusternor)
nstart <- 5
kmeansplusplus <- KmeansPP(wine.stand, 3, nstart=nstart)
# Cluster size:
kmeansplusplus[["size"]][1] 86 42 50# Clusters:
kmeansplusplus[["cluster"]] [1] 2 3 2 2 1 2 2 2 3 3 2 3 3 2 2 2 2 2 2 2 2 1 3 3 2 1 3 3 2 3 2 2 3 2 2 2 3
[38] 3 3 2 2 3 2 3 3 2 2 2 2 2 3 2 2 2 2 2 2 3 2 3 3 3 3 3 1 3 3 3 3 2 1 1 1 2
[75] 3 3 3 1 2 1 3 3 1 1 3 3 1 1 1 1 1 1 1 3 3 2 1 3 3 1 3 3 1 3 3 1 3 1 3 1 1
[112] 1 1 1 1 1 3 1 3 1 1 1 1 1 1 1 1 1 1 1 3 1 1 1 3 1 1 1 1 1 1 1 1 1 3 1 1 1
[149] 1 1 1 1 1 1 1 1 1 1 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1Cluster Fuzzy
Esse seguimento é uma tradução desse site: https://www.datanovia.com/en/lessons/fuzzy-clustering-essentials/
O Cluster Fuzzy também é conhecido como método flexível. As abordagens de cluster padrão produzem partições (K-means, PAM), nas quais cada observação pertence a apenas um cluster. Isso é conhecido como clustering rígido. Em outras palavras, se o josé foi selecionado para o cluster 1, ele não pode pertencer ao cluster 2. Ele não pode estar simultaneamente no cluster 1 e 2. Na linguagem estatística isso se chama mutualmente excludente.
No cluster Fuzzy, os itens podem ser membros de mais de um cluster ao mesmo tempo. Cada item possui um conjunto de coeficientes de associação que correspondem ao grau de presença a um determinado cluster. O método fuzzy c-means (FCM) é o algoritmo de agrupamento Fuzzy mais popular.
A função fanny() [do pacote cluster] pode ser usada para calcular o cluster Fuzzy. FANNY está relacionado ao acrônimo de Fuzzy Analysis clustering.
Um formato simplificado do cluster Fuzzy FANNY é:
fanny(x, k, metric = "euclidean", stand = FALSE)Onde:
x: uma matriz ou data.frame ou matriz de dissimilaridade.
k: o número desejado de clusters a serem gerados.
metric: métrica para calcular dissimilaridades entre observações.
stand: Se TRUE, as variáveis são padronizadas antes de calcular as diferenças.
A função fanny() retorna um objeto incluindo os seguintes componentes:
* Membership: matriz que contém o grau em que cada observação pertence a um determinado *cluster*. Os nomes das colunas são os clusters e as linhas são observações.
* Coeficiente: o coeficiente de partição F(k) de Dunn do cluster, em que k é o número de clusters. F(k) é a soma de todos os coeficientes de associação ao quadrado, divididos pelo número de observações. Seu valor está entre 1/k e 1. A forma normalizada do coeficiente também é fornecida. É definido como (F(k) −1/k) / (1 −1/k), e varia entre 0 e 1. Um valor baixo do coeficiente de Dunn indica um agrupamento muito confuso, enquanto um valor próximo a 1 indica um agrupamento quase nítido.
* Clustering: o vetor de clustering que contém o agrupamento mais nítido de observações.Por exemplo, o código do R abaixo aplica clustering nebuloso (cluster Fuzzy) no banco de dados USArrests:
library(cluster)
df <- scale(USArrests) # Standardize the data
res.fanny <- fanny(df, 2) # Compute fuzzy clustering with k = 2Os diferentes componentes podem ser extraídos usando o código abaixo:
head(res.fanny$membership, 3) # Membership coefficients [,1] [,2]
Alabama 0.6641977 0.3358023
Alaska 0.6098062 0.3901938
Arizona 0.6862278 0.3137722res.fanny$coeff # Dunn's partition coefficientdunn_coeff normalized
0.5547365 0.1094731 head(res.fanny$clustering) # Observation groups Alabama Alaska Arizona Arkansas California Colorado
1 1 1 2 1 1 Para visualizar grupos de observação, use a função fviz_cluster() [pacote fatoextra]:
library(factoextra)
fviz_cluster(res.fanny, ellipse.type = "norm", repel = TRUE,
palette = "jco", ggtheme = theme_minimal(),
legend = "right")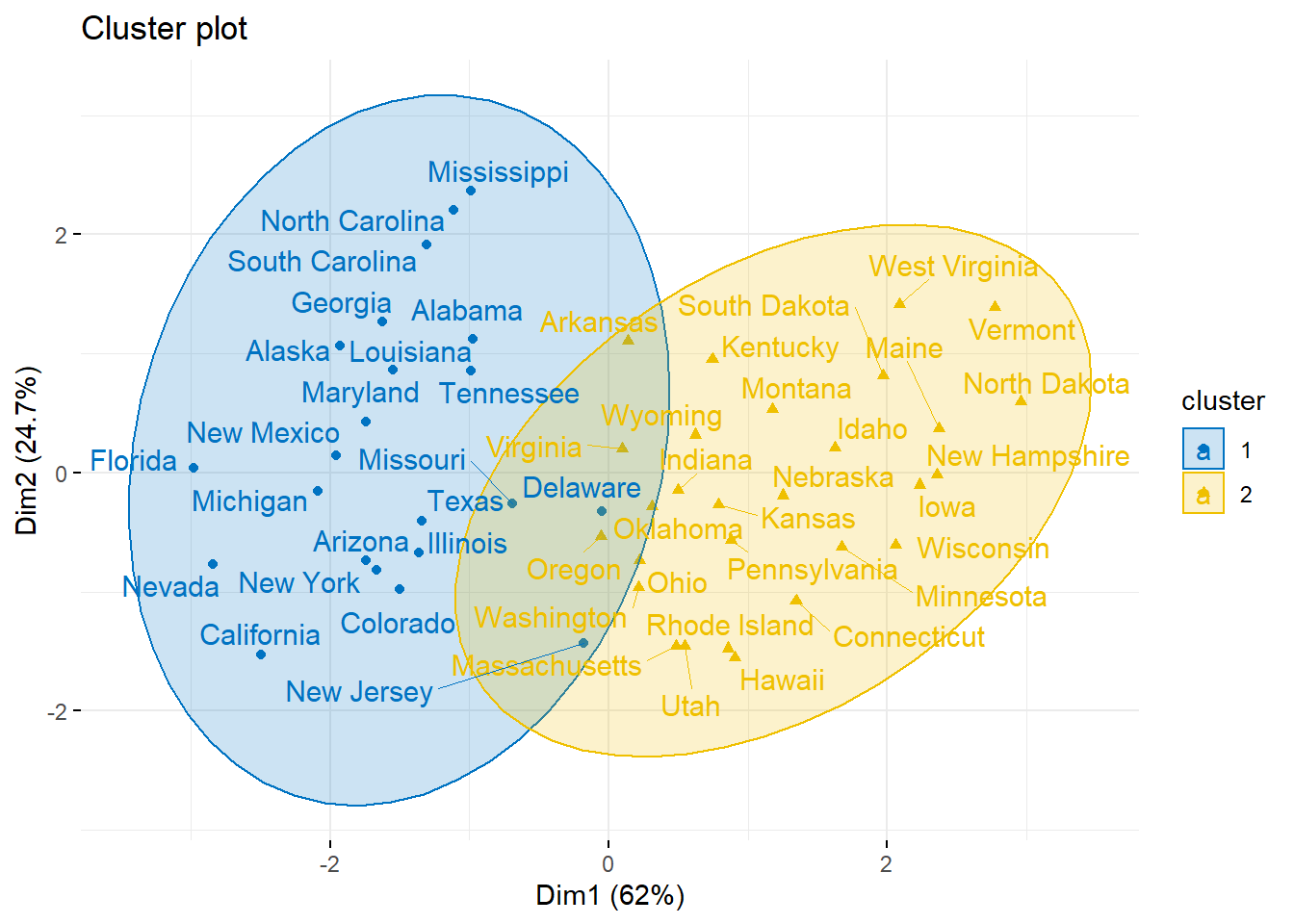
Para avaliar a qualidade do ajuste dos cluster aos dados, podemos usar o método silhouette (vou apresentá-lo abaixo) da seguinte maneira:
fviz_silhouette(res.fanny, palette = "jco",
ggtheme = theme_minimal()) cluster size ave.sil.width
1 1 22 0.32
2 2 28 0.44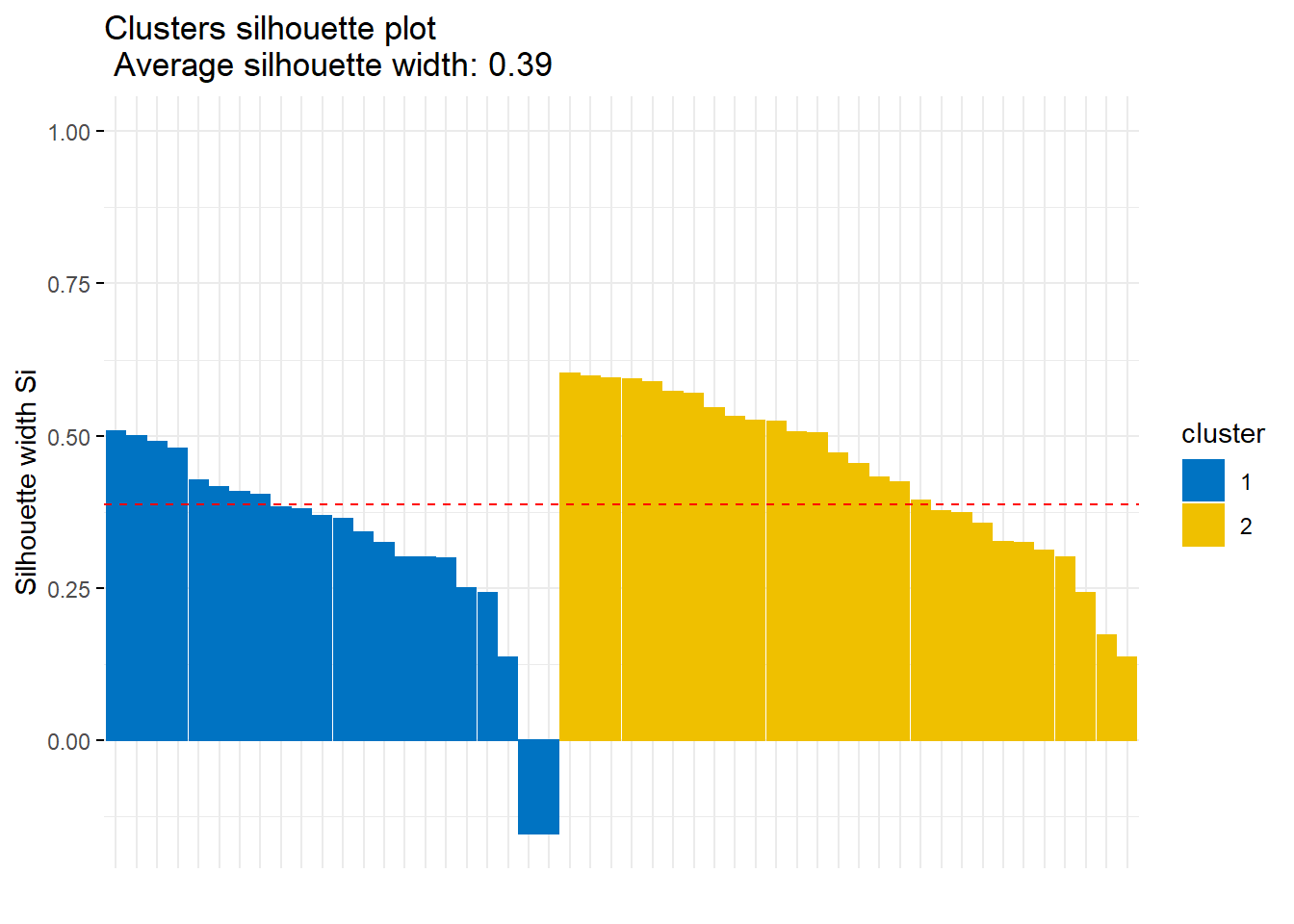
O cluster Fuzzy é uma alternativa ao k-means, em que cada ponto de dados possui coeficiente de associação para cada cluster.
Cluster para variáveis categóricas
Distância de Gower
O conceito de distância de Gower é simples. Para cada tipo de variável, uma métrica de distância específica que funciona para esse tipo/classe é usada e dimensionada para ficar entre 0 e 1. Em seguida, uma combinação linear usando pesos (uma média ponderada) é calculada para criar a matriz de distância final. As métricas usadas para cada tipo de dados são descritas abaixo:
- Quantitativa (intervalar): distância de Manhattan com intervalo normalizado.
- Ordinal: a variável é classificada primeiro, depois a distância de Manhattan é usada com um ajuste especial para os empates.
- Nominal: As k categorias da variável são primeiro convertidas em k colunas binárias e, em seguida, o coeficiente dice é utilizado.
Abaixo, vemos que a distância de Gower pode ser calculada em uma linha usando a função daisy.
# To standarize the variables
type<-wine$Type
wine.stand2<-merge(wine.stand,type,by="row.names",all.x=TRUE)
gower_dist <- daisy(wine.stand2, metric = "gower",type = list(ordratio = 1))Partitioning Around Medoids - PAM
A partição PAM é um procedimento de cluster iterativo com as seguintes etapas:
- Escolher k entidades aleatórias para se tornar os medoids.
- Atribuir todas as entidades ao seu medoids mais próximo (usando uma matriz de distância).
- Para cada cluster, identificar a observação que produziria a menor distância média se fosse redesignada como medoid Nesse caso, faça dessa observação o novo medoid.
- Se pelo menos um medoid tiver sido alterado, retorne à etapa 2. Caso contrário, termine o algoritmo.
pam_fit <- pam(gower_dist,diss = TRUE,k = 3)
pam_fit Medoids:
ID
[1,] 109 109
[2,] 164 164
[3,] 56 56
Clustering vector:
[1] 1 1 2 2 2 2 2 2 2 2 2 2 1 2 2 2 2 2 2 2 2 2 3 1 2 2 1 2 2 2 2 2 2 2 1 2 3
[38] 3 3 3 3 3 3 3 3 1 3 3 3 3 3 3 3 3 3 3 1 3 3 3 3 3 3 3 3 3 3 1 3 3 3 3 3 3
[75] 3 3 3 3 1 3 3 3 3 3 3 3 3 3 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
[112] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 1 2 2
[149] 2 2 2 2 2 2 2 2 1 2 2 2 2 2 2 2 2 2 2 1 2 2 2 2 2 2 2 2 2 2
Objective function:
build swap
0.1278866 0.1224580
Available components:
[1] "medoids" "id.med" "clustering" "objective" "isolation"
[6] "clusinfo" "silinfo" "diss" "call" Density-based clustering
Uma outra abordagem para gerar agrupamentos é o Density-based clustering. A seguir mostro um pouco do código para gerar esse tipo de análise. A fonte desse código é prof. Bharatendra Rai.
data("iris")
str(iris)'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...new <- iris[,-5]
# Installing packages
#devtools::install_github("kassambara/factoextra")
library(fpc)
library(dbscan)
library(factoextra)
# Obtaining optimal eps value
kNNdistplot(new, k=3)
abline(h = 0.45, lty=2)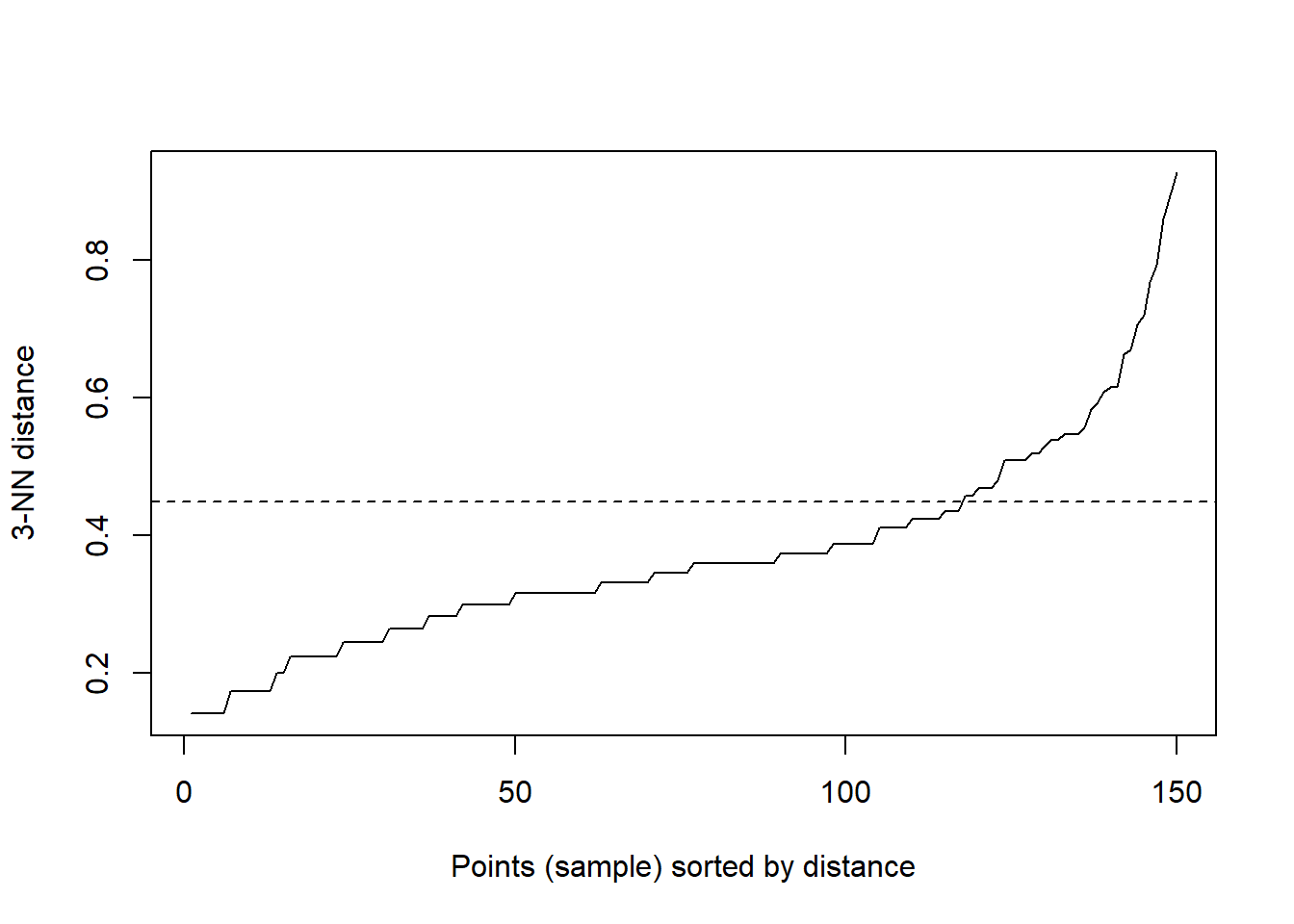
# Density-based clustering with fpc & dbscan
set.seed(12345)
f <- fpc::dbscan(new, eps=0.45, MinPts = 2)
d <- dbscan::dbscan(new, 0.45, 2)
# Cluster visualization
fviz_cluster(d, new, geom = "point")
t-SNE plot
t-distributed Stochastic Neighbor Embedding (t-SNE) é um algoritmo de machine learning para visualização desenvolvido por Laurens van der Maaten and Geoffrey Hinton. É uma técnica de redução de dimensionalidade não linear, adequada para incorporar dados de alta dimensão para visualização dos dados.
library(Rtsne)
tsne_obj <- Rtsne(gower_dist, is_distance = TRUE)
tsne_data <- tsne_obj$Y %>%
data.frame() %>%
setNames(c("X", "Y")) %>%
mutate(cluster = factor(pam_fit$clustering))
library(ggplot2)
ggplot(aes(x = X, y = Y), data = tsne_data) +
geom_point(aes(color = cluster))
# Multi-Dimensional Reduction and Visualisation with t-SNE
# https://datascienceplus.com/multi-dimensional-reduction-and-visualisation-with-t-sne/Como determinar o número ótimo de clusters:
Para determinar o número de clusteres, use o código:
factoextra::fviz_nbclust()Existem três grandes métodos para avaliar o número de cluster. 1. Método do cotovelo 2. Método de silhueta (Average Silhouette Method) 3. Método Gap (Gap Statistic Method)
Método do cotovelo
Para executar o método do cotovelo (Elbow Method), basta executar o seguite código:
fviz_nbclust(df, FUN = hcut, method = "wss")Método de silhueta (Average Silhouette Method)
Para utilizar o método da silhueta, seguimos um processo parecido.
fviz_nbclust(df, FUN = hcut, method = "silhouette")Método Gap (Gap Statistic Method). Mind de Gap
Para utilizar esse processo, basta executar:
gap_stat <- clusGap(df, FUN = hcut, nstart = 25, K.max = 10, B = 50)
fviz_gap_stat(gap_stat)library("factoextra")
set.seed(12345)
fviz_nbclust(wine.stand, kmeans, method = "wss")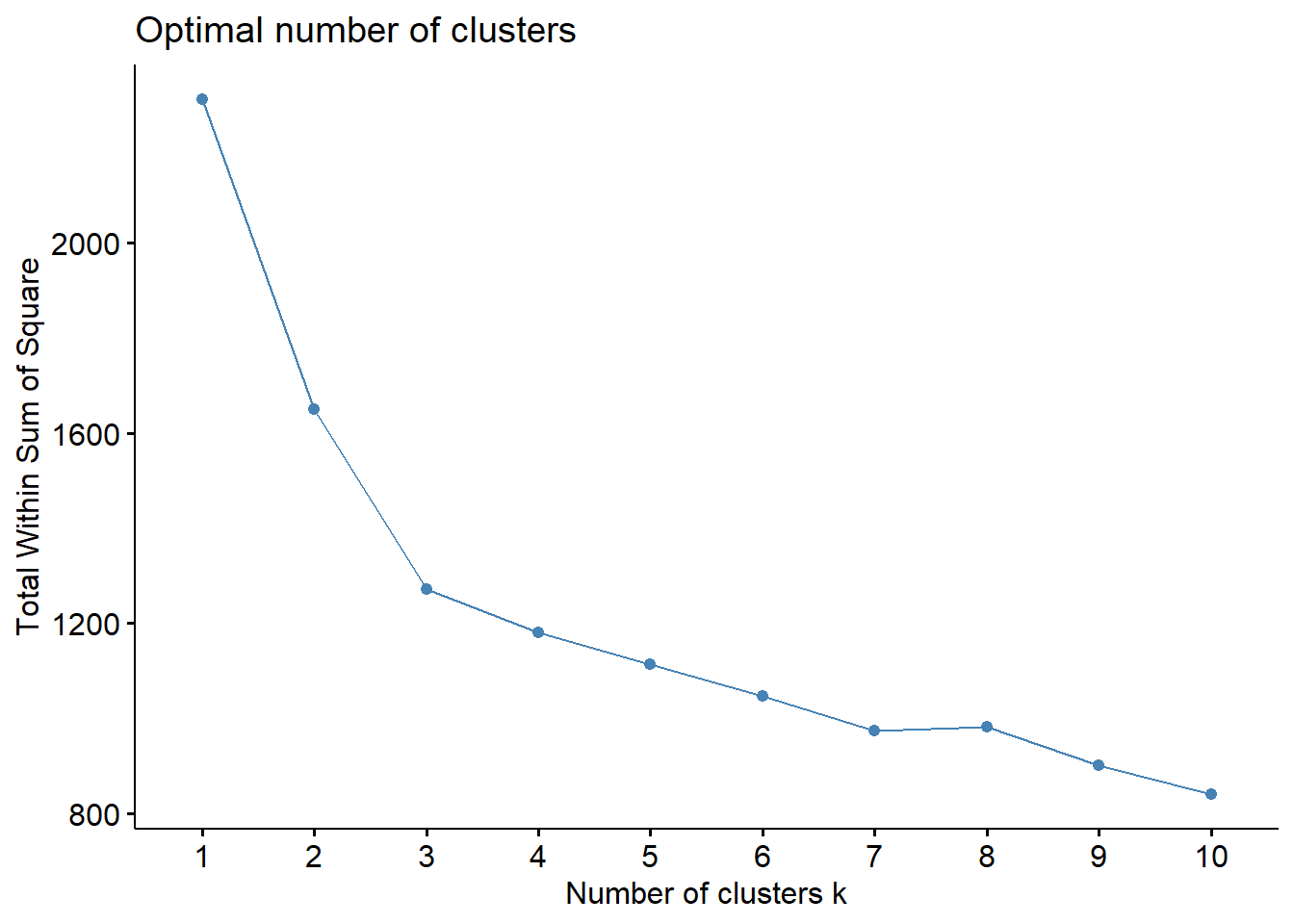
fviz_nbclust(wine.stand, kmeans, method = "silhouette")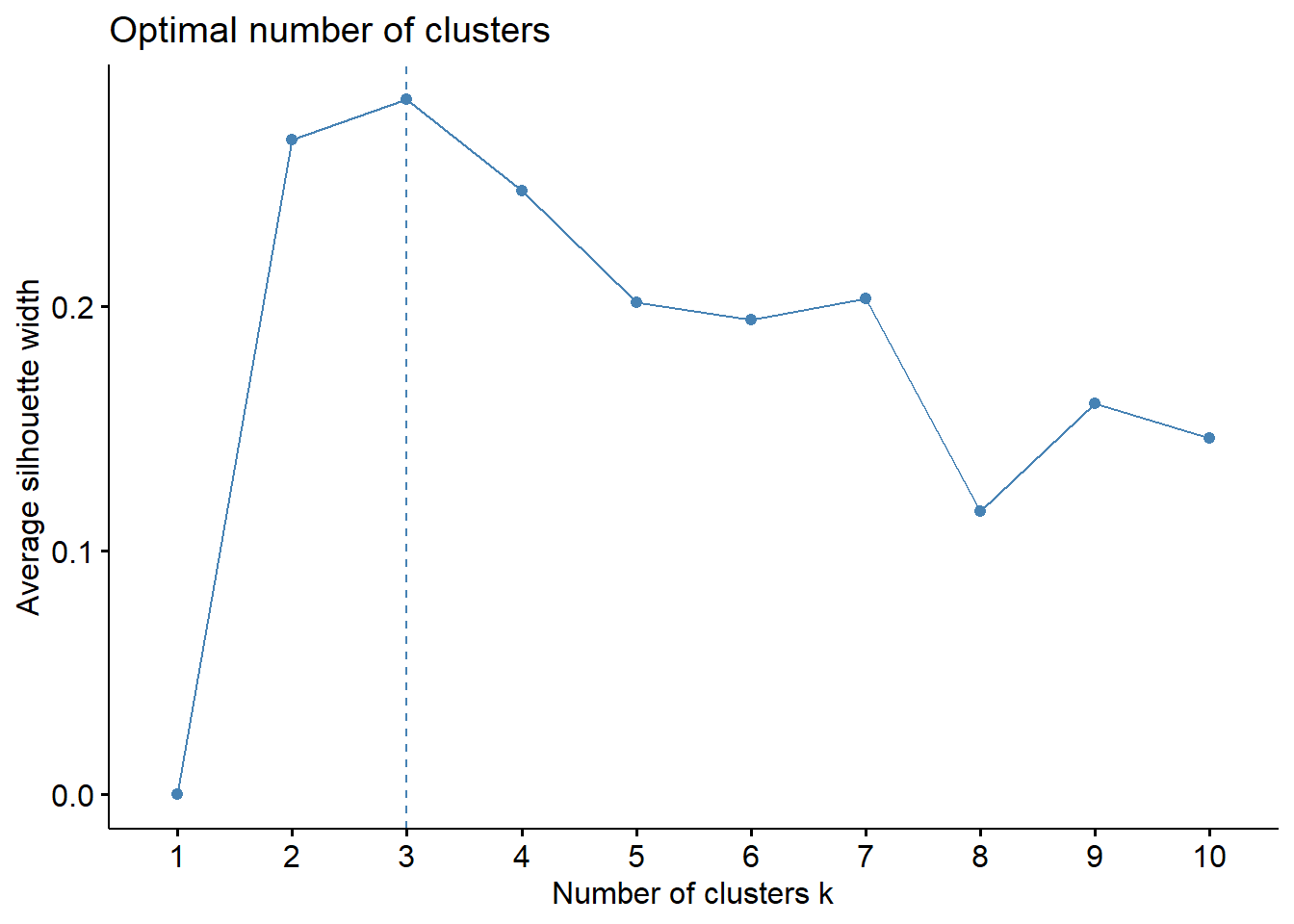
fviz_nbclust(wine.stand, kmeans, method = "gap_stat")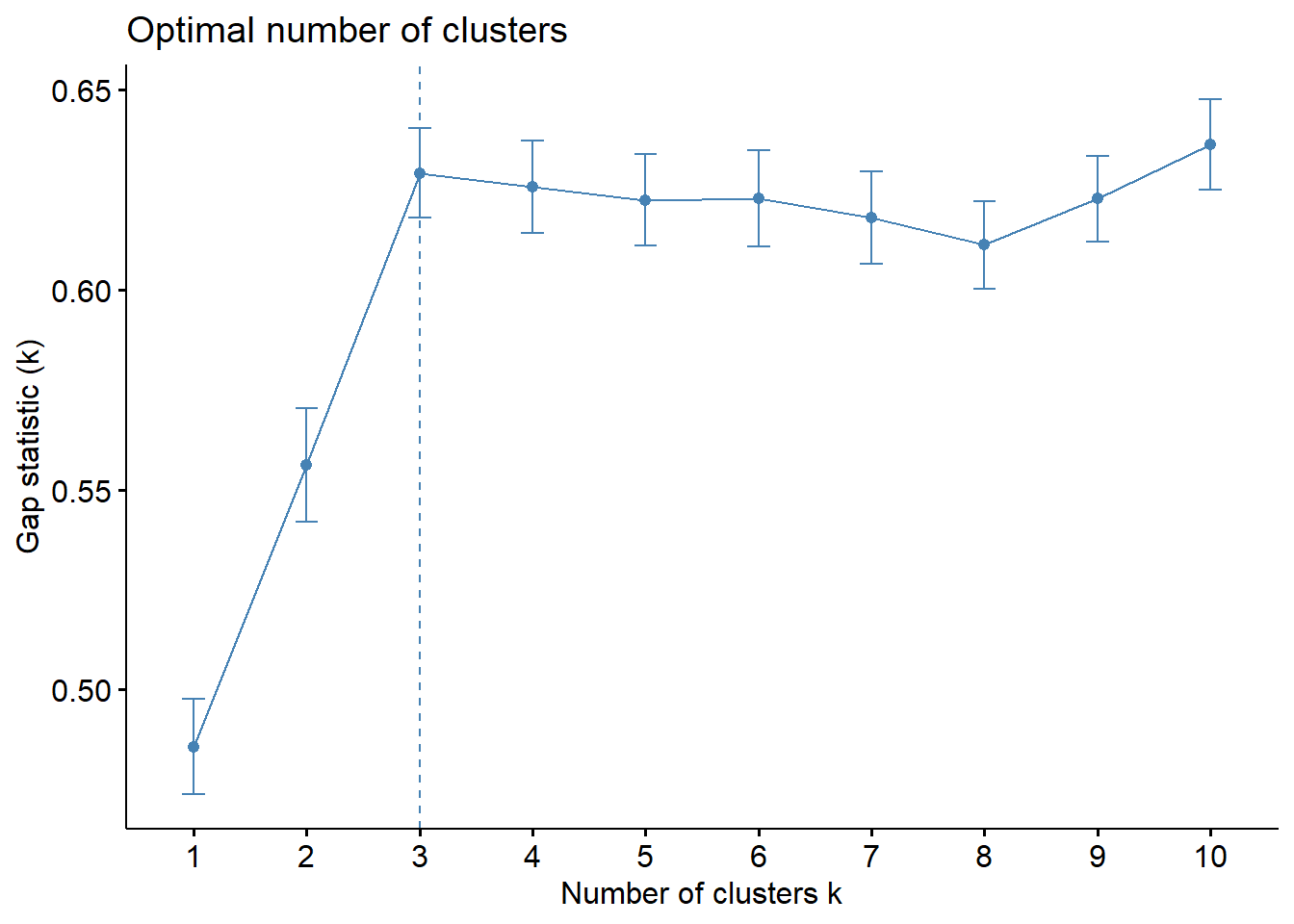
#gap_stat <- clusGap(wine.stand, FUN = kmeans, nstart = 25,K.max = 10, B = 50)
#fviz_gap_stat(gap_stat)Parece que três grupos (k = 3) é a melhor quantidade de grupos pelos três os gráficos.
Comentários adicionais
A análise de cluster pode ser uma ferramenta útil para análise de dados. No entanto, existem vários problemas que surgem na execução de cluster. Por exemplo, no caso de cluster hierárquico, precisamos nos preocupar com:
- Que medida de dissimilaridade deve ser usada?
- Que tipo de ligação deve ser usada?
- Onde devemos cortar o dendograma para obter clusteres?
Cada uma dessas decisões tem um forte impacto nos resultados obtidos. Na prática, tentamos várias opções diferentes e procuramos aquela com a solução interpretável. Com esses métodos, não há uma única resposta correta - qualquer solução que exponha alguns aspectos interessantes dos dados deve ser considerada. Outras informações na bibliografia.
Parte dessa publicação é uma tradução livre dos seguintes sites: https://www.datanovia.com/en/blog/types-of-clustering-methods-overview-and-quick-start-r-code/ e https://uc-r.github.io/hc_clustering.
Se você usar o código ou as informações deste guia em um trabalho publicado, solicito que cite-o como uma fonte nas referências bibliográficas.
DUTT-ROSS,Steven Métodos de Análise de Cluster. Rio de Janeiro. 2020. mimeo. Disponível em: https://blog.metodosquantitativos.com/cluster/
Outras referências desse texto.
1. https://www.datanovia.com/en/blog/types-of-clustering-methods-overview-and-quick-start-r-code/
2. https://uc-r.github.io/kmeans_clustering
3. https://data-flair.training/blogs/clustering-in-r-tutorial/
4. http://dpmartin42.github.io/posts/r/cluster-mixed-types
Para saber sobre outras funções no e no Python, você pode acessar o meu blog https://blog.metodosquantitativos.com/ ou o meu site pessoal https://steven.metodosquantitativos.com/
